以下14行目のコードでXX、YY、ZZをそれぞれ(50,50)から(2500,)に変換しているようなのですが、これはdecision_functionがこの形式しか受け付けないからでしょうか。
np.c_[XX.ravel(),YY.ravel(),ZZ.ravel()] ですが、
XX, YY, ZZ はそれぞれ (50, 50) の2次元配列であり、ndarray.ravel() でそれぞれ (2500,) の1次元配列に変更しています。numpy.c_[XX.ravel(),YY.ravel(),ZZ.ravel()] で3つの1次元配列を横方向に結合しているので、(2500, 3) の2次元配列になります。
この (2500, 3) の2次元配列を decision_function() に渡しています。
また、decision_functionは何をしているのでしょうか。fitと同じように決定境界を学習しているのでしょうか。
学習自体は fit() で完了しています。
decision_function() は、点を与えると、SVM が学習した分離超平面からの距離を返します。
sklearn.svm.LinearSVC.decision_function
以下15行目のコードでXX,YYの右にdec.reshape(XX.shape)が入るのはなぜでしょうか。これは(2500,)のdecを(50,50)にしているようですが、なぜXX,YY,ZZではないのでしょうか。
dec.reshape(XX.shape) としているのは、contourf() の仕様上、dec の形状 (2500,) を XX, YY に合わせて (50, 50) にしなければいけないためです。
コードの流れ解説
線形分離できない2次元のデータが与えられたとします。
python
1import matplotlib.pyplot as plt
2import numpy as np
3from matplotlib.colors import ListedColormap
4from mpl_toolkits.mplot3d import Axes3D
5from sklearn.datasets import make_blobs
6from sklearn.svm import LinearSVC
7
8# 2次元のデータを作成する。
9X, y = make_blobs(centers=4, random_state=8)
10y = y % 2
11
12fig, ax = plt.subplots()
13class_colors = ListedColormap(["g", "k"])
14ax.scatter(X[:, 0], X[:, 1], c=y, cmap=class_colors)
15ax.set_xlabel("Feature 0")
16ax.set_ylabel("Feature 1")
17plt.show()
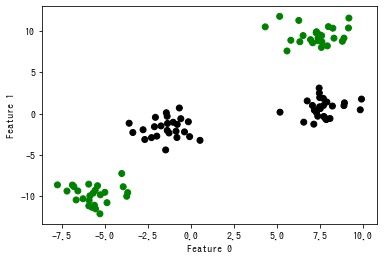
このままでは線形分離できないので、Φ: (x, y) → (x, y, y^2) という関数で3次元空間に射影します。3次元空間上に射影したことで線形分離可能になったので、線形 SVM で学習できます。
python
1X_new = np.hstack([X, X[:, 1:] ** 2])
2
3fig = plt.figure(figsize=(7, 7))
4ax = fig.add_subplot(111, projection="3d")
5ax.scatter(X_new[:, 0], X_new[:, 1], X_new[:, 2], c=y, cmap=class_colors)
6ax.view_init(30, 30)
7ax.set_xlabel("Feature 0")
8ax.set_ylabel("Feature 1")
9plt.show()

fit() で学習します。
python
1svc = LinearSVC().fit(X_new, y)
射影した空間 {(x, y, y^2)|x, y∈ℝ} 上の各点と分類超平面との距離を decision_function() で計算します。
python
1# 射影した空間上の点を作成する。
2xs = np.linspace(X_new[:, 0].min() - 2, X_new[:, 0].max() + 2, 50)
3ys = np.linspace(X_new[:, 1].min() - 2, X_new[:, 1].max() + 2, 50)
4XX, YY = np.meshgrid(xs, ys)
5ZZ = YY ** 2
6
7# 作成した点と分類超平面との距離を計算する。
8dist = svc.decision_function(np.c_[XX.ravel(), YY.ravel(), ZZ.ravel()])
9
10# SVM の超平面を計算する。
11def hyper_plane(svc, XX, YY):
12 a1, a2, a3 = svc.coef_[0]
13 b = svc.intercept_[0]
14 return (-b - a1 * XX - a2 * YY) / a3
15
16
17plane = hyper_plane(svc, XX, YY)
18
19
20# 可視化する。
21fig = plt.figure(figsize=(9, 7))
22ax = fig.add_subplot(111, projection="3d")
23# 学習データの点を可視化する。
24ax.scatter(X_new[:, 0], X_new[:, 1], X_new[:, 2], c=y, cmap=class_colors)
25# 射影した空間上の各点と分類超平面との距離を可視化する。
26sc = ax.scatter(XX, YY, ZZ, c=dist, cmap="bwr", vmin=-15, vmax=15)
27# 分類超平面を可視化する。
28ax.plot_surface(XX, YY, plane, alpha=0.5)
29ax.view_init(30, 30)
30ax.set_xlabel("Feature 0")
31ax.set_ylabel("Feature 1")
32fig.colorbar(sc)
33plt.show()
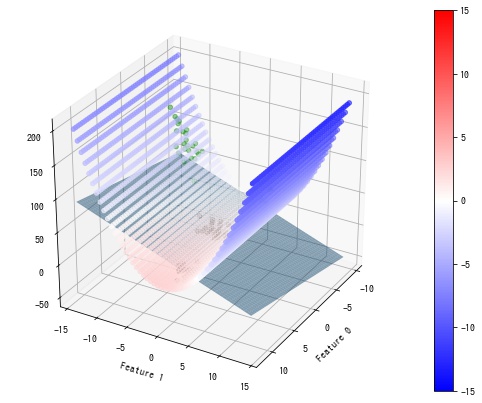
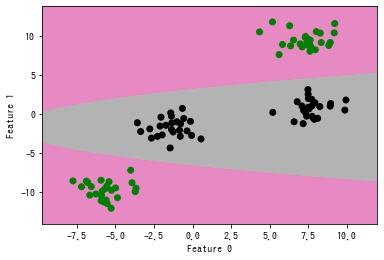
{(x, y)|decision_function(x, y, y^2) = 0, x, y∈ℝ} が元の2次元空間において、決定境界となります。
なので、decision_function(x, y, y^2) = 0 である等高線を contourf() で描画します。
python
1fig, ax = plt.subplots()
2class_colors = ListedColormap(["g", "k"])
3# 決定境界を可視化する。
4ax.contourf(
5 XX, YY, dist.reshape(XX.shape), levels=[dist.min(), 0, dist.max()], cmap="Set2"
6)
7# データを可視化する。
8ax.scatter(X[:, 0], X[:, 1], c=y, cmap=class_colors)
9plt.xlabel("Feature 0")
10plt.ylabel("Feature 1")
11plt.show()








2020/05/23 14:45
2020/05/23 16:19
2020/05/24 07:07
2020/05/24 07:19 編集
2020/05/24 11:48